Comme on a pu le voir par exemple dans le précédent post, l’aggrégation est souvent utilisée en analyse de données. Il est donc intéressant de comparer les performances des différentes options que propose R de ce point de vue. Des benchmarks comparant data.table, dplyr et la librairie pandas de python sur différentes tailles de tables ont déjà été faits, vous pouvez les trouver sur cette page github. On propose ici quelques tests comparatifs complémentaires sur un cas d’un calcul simple à partir d’un groupement d’une base fictive de nbrow lignes appartenant à nbgpe groupes. La fonction s’applique à deux variables numériques x et y, la première étant une variable aléatoire et la seconde un entier dont on fait varier le nombre de modalités. On teste l’instruction suivante :
Pour dplyr
datatib %>% group_by(y) %>% summarise(x = mean(x))Pour data.table
dataDT[, .(x = mean(x)), by = .(y = y)]Pour base R
tap <- tapply(test$x, test$y, mean)
data.frame(x = tap, y = names(tap))Notons que dans ce dernier cas, on ajoute une étape pour transformer l’output en dataframe. On aurait aussi pu utiliser la fonction aggregate qui permet cela.
Il n’y a plus qu’à tester! On propose des tests sur 10 000, 100 000 et 1 million de lignes avec à chaque fois peu (1/1000e du nombre de lignes) ou beaucoup (la moitié du nombre de lignes) de groupes. On fait un tableau récapitulatif des différents graphiques issus de la fonction autoplot de ggplot2 qui sort joliment les résultats de microbenchmark (on regroupe ces graphiques à l’aide du package gridExtra). Les graphiques représentent la distribution du temps d’exécution des 10 occurences testées par méthode.

Le premier constat est que la méthode data.table est toujours plus rapide que les alternatives testées. Le second est que R base concurrence dplyr dans tous les cas où le nombre de groupes sur lesquels il faut agréger est petit. Au contraire, la fonction tapply est largement en dessous des performances des deux autres options quand le nombre de groupes est élevé.
Le changement d’échelle du graphique à chaque hypothèse testée est trompeur mais ne doit pas cacher que dans le cas d’une table à 1 million de lignes et 50 000 groupes, si l’option dplyr fait largement mieux que R base, elle est aussi plus surpassée que jamais par data.table. Regardons le graphique de résultats de cette hypothèse pour mieux s’en rendre compte :
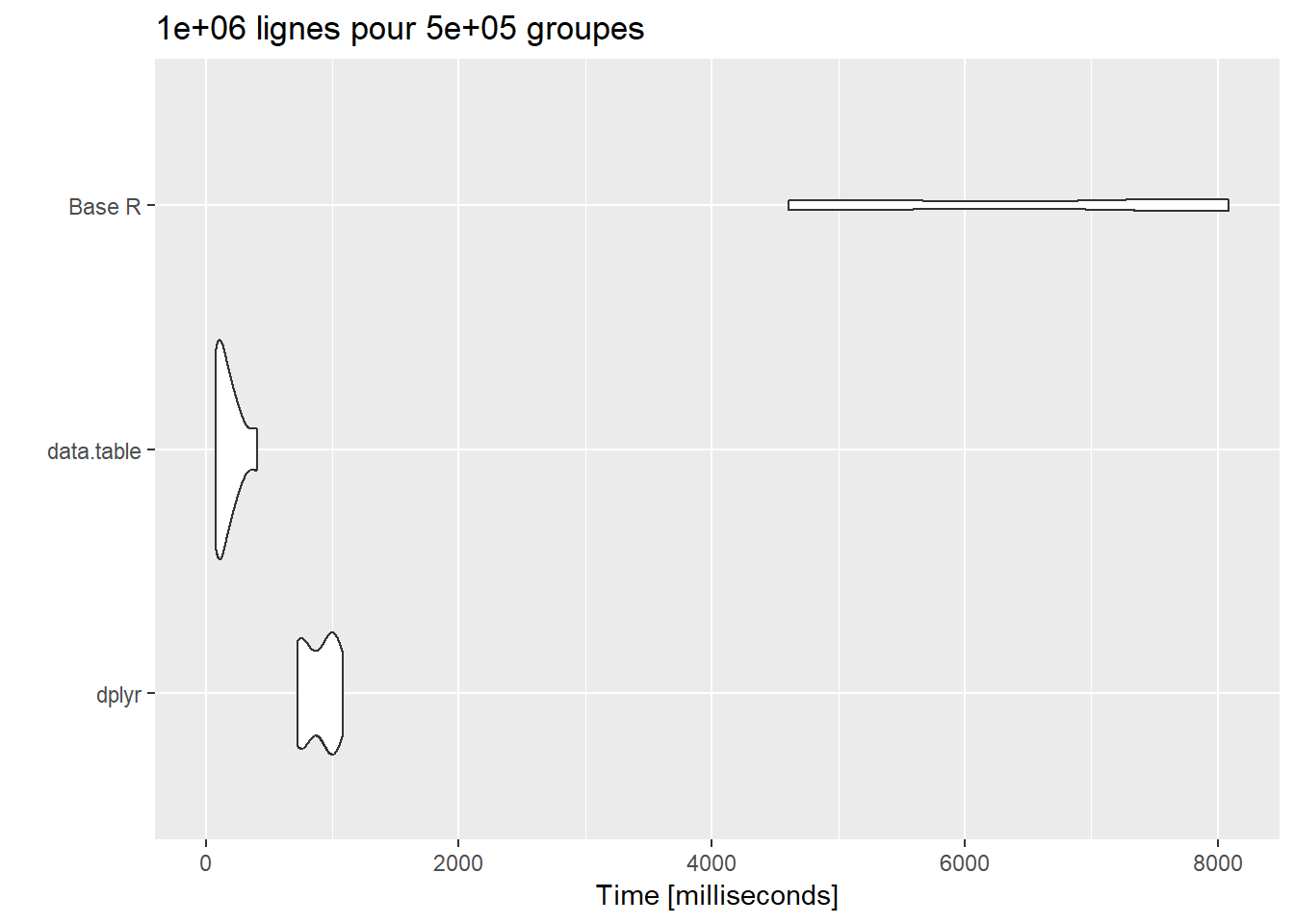
Les temps d’exécution de data.table se situent autour de 100 millisecondes alors que ceux de dplyr sont autour de 800 millisecondes.
Ces tests confirment ceux cités en introduction de ce post. Ils montrent l’intérêt d’utiliser data.table dans le cas d’instructions agrégées si l’on souhaite optimiser le temps d’exécution de son script et/ou si l’on connaît des difficultés à gérer des tables volumineuses. Ils montrent aussi que dplyr reste une option crédible et très compétitive notamment par rapport aux fonctions de base R.